Do gene à proteína

Pré-requisito: conhecer a estrutura do DNA – acesse aqui
Os genes fornecem as instruções para a produção de proteínas específicas. No entanto, um gene não sintetiza uma proteína diretamente. A conexão entre o DNA e a síntese de proteínas é o ácido nucleico RNA. O RNA é quimicamente similar ao DNA, com a diferença de conter uma ribose, em vez de uma desoxirribose, como açúcar, e por possuir a base nitrogenada uracila, em vez da timina
Dessa forma, cada nucleotídeo ao longo de uma fita de DNA apresenta A, G, C ou T como base, e cada nucleotídeo ao longo de uma fita de RNA apresenta A, G, C ou U como base. A molécula de RNA geralmente é composta por uma fita simples.
Os genes geralmente são compostos por centenas ou milhares de nucleotídeos, onde cada gene apresenta uma sequência específica de nucleotídeos. Cada proteína também apresenta uma série de monômeros arranjados em ordem linear específica (a sequência primária de uma proteína); mas os seus monômeros são os aminoácidos.
Dessa forma, os ácidos nucleicos e as proteínas contêm informações escritas em duas linguagens químicas distintas. O caminho do DNA até as proteínas exige duas etapas principais: transcrição e tradução.
Pense assim: o DNA como um caderno de receitas da célula
Imagine que o núcleo da célula é uma cozinha-chefe super protegida, onde está guardado um caderno muito valioso: o DNA. Esse caderno contém todas as receitas que a célula precisa para funcionar — e cada receita corresponde a um gene, que traz as instruções para produzir uma proteína específica (como uma enzima, por exemplo). Entretanto, esse caderno não pode sair do núcleo, porque é muito importante e precisa ser bem protegido.
Entra em cena o RNA mensageiro (RNAm), que funciona como um ajudante de cozinha. Ele entra na cozinha, copia uma receita (gene) à mão, e sai correndo para a área de produção da célula — o citoplasma, onde ficam os ribossomos (as “fábricas” de proteínas). Esse processo de copiar a receita do DNA para o RNAm é chamado de transcrição — como se estivéssemos transcrevendo um texto.
Depois, no citoplasma, os ribossomos leem a receita que o RNAm trouxe e seguem as instruções para montar a proteína, juntando os ingredientes certos (os aminoácidos) na ordem correta. Esse segundo processo, de ler a receita e produzir a proteína, é chamado de tradução — como se estivéssemos traduzindo a linguagem dos nucleotídeos (letras do RNA) para a linguagem das proteínas (aminoácidos).
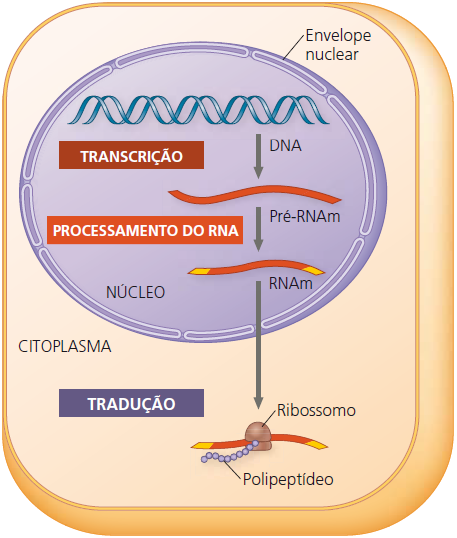
A transcrição
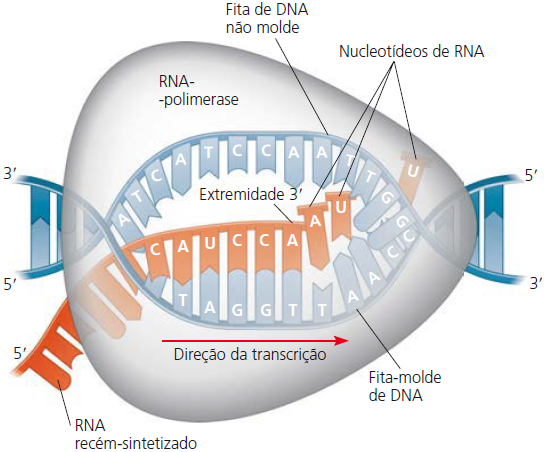
Transcrição é a síntese de RNA utilizando informações contidas no DNA. Ambos os ácidos nucleicos utilizam a mesma linguagem, e a informação é simplesmente transcrita, ou “copiada”, do DNA para o RNA. Esse tipo de molécula de RNA é chamada de RNA mensageiro (RNAm), pois carrega a mensagem genética do DNA até a maquinaria de síntese proteica da célula.
Para cada gene, apenas uma das duas fitas de DNA é transcrita. Essa fita é chamada de fita-molde, pois fornece o padrão, ou molde, para a sequência de nucleotídeos no transcrito de RNA. Para um gene em específico, a mesma cadeia é utilizada como molde a cada vez que o gene é transcrito. Para outros genes na mesma molécula de DNA, no entanto, a cadeia oposta pode ser a cadeia molde.
A molécula de RNAm é complementar, e não idêntica, ao DNA molde, pois os nucleotídeos da molécula de RNA são adicionadas sobre a fita molde de acordo com a regra de pareamento de bases Os pares de bases são similares àqueles formados durante a replicação do DNA, exceto pela presença de U (o substituto de T na molécula de RNA) pareando com A.
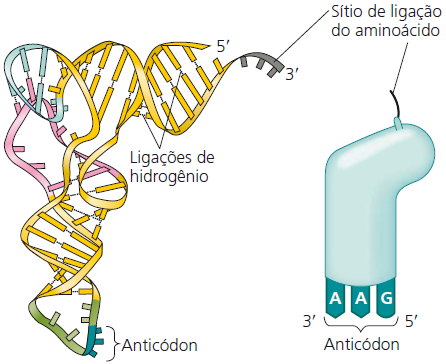
Estrutura e função do RNA transportador
A chave para a tradução da mensagem genética em uma sequência específica de aminoácidos é o fato de que cada molécula de RNAt traduz um códon específico de RNAm em um determinado aminoácido. Isso é possível, pois o RNAt possui um aminoácido específico em uma das duas extremidades, enquanto a outra extremidade corresponde a uma trinca de nucleotídeos (anticódon) que pode se parear com o códon complementar na cadeia de RNAm.
A tradução
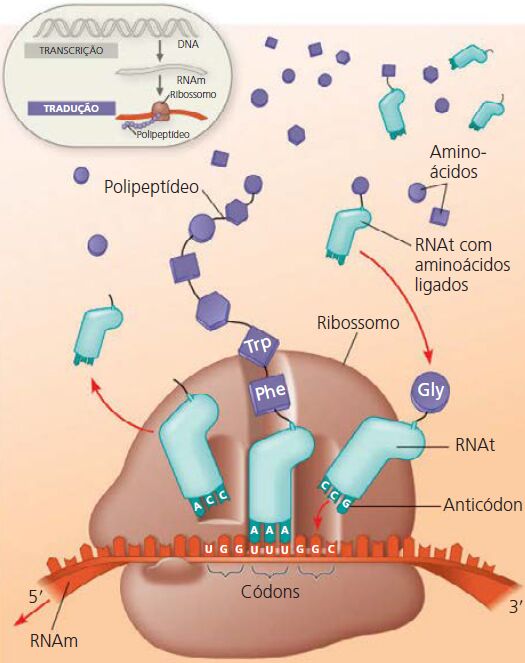
Tradução é a síntese de um polipeptídeo utilizando a informação contida no RNAm. Durante essa etapa, ocorre uma mudança na linguagem: a célula precisa traduzir a sequência de nucleotídeos da molécula de RNAm em uma sequência de aminoácidos do polipeptídeo. Os locais de tradução são os ribossomos, complexos moleculares que facilitam a adição ordenada de aminoácidos às cadeias polipeptídicas.
Códon: a trinca de nucleotídeos
Se cada base do DNA fosse responsável por codificar diretamente um aminoácido, só teríamos quatro aminoácidos diferentes — um para cada tipo de base (A, T, C ou G). Mas como o nosso corpo precisa produzir 20 aminoácidos diferentes, seria necessário usar combinações de bases.
E se tentássemos usar duplas de bases (como “AG” ou “GT”) para formar os códigos? Como existem 4 bases possíveis e duas posições por combinação, teríamos:
- 4 × 4 = 16 combinações possíveis.
Isso ainda não seria suficiente para representar os 20 aminoácidos. Por isso, o código genético usa combinações de três bases (trincas), chamadas de códons, o que gera:
- 4 × 4 × 4 = 64 combinações possíveis — mais do que o necessário, o que permite repetições e códigos de parada.
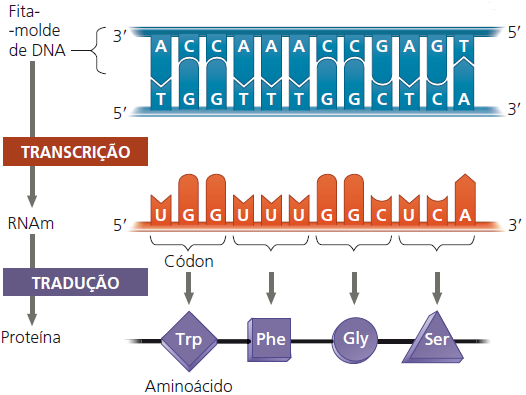
O código de trincas. Para cada gene, uma das fitas de DNA serve de molde para a transcrição de moléculas de RNA, assim como RNAm. A regra de pareamento de bases para a síntese de DNA também guia a transcrição, mas a uracila (U) substitui
a timina (T) no RNA. Durante a tradução, a molécula de RNAm é lida como uma sequência de trincas de nucleotídeos, chamadas de códons. Cada códon especifica um aminoácido que será adicionado à cadeia polipeptídica em crescimento. A molécula de RNAm é lida na direção 5′ 🠖 3′.
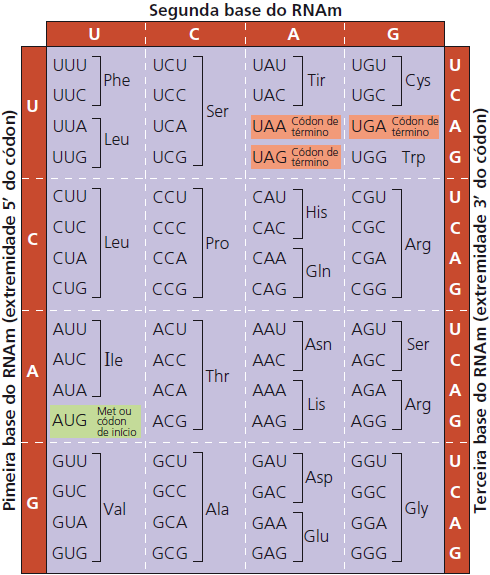
61 dos 64 códons codificam aminoácidos. Os três códons que não correspondem a aminoácidos são sinais de “parada” ou códons de término, marcando o final da tradução. Observe que o códon AUG tem função dupla: codifica o aminoácido metionina (Met) e também atua como sinal de início ou códon de início. As mensagens genéticas iniciam com o códon AUG no RNAm, que sinaliza para a maquinaria de síntese de proteínas que a tradução do RNAm deve começar nesta posição.
Há redundância no código genético, mas não ambiguidade. Por exemplo, apesar de os dois códons GAA e GAG codificarem o ácido glutâmico (redundância), nenhum deles especifica outro aminoácido (ausência de ambiguidade). A redundância no código não é aleatória. Em diversos casos, os códons sinônimos para um aminoácido em particular diferem apenas na terceira base da trinca.
Chamamos de degenerado esse fenômeno de redundância no código genético, no qual mais de um códon pode codificar o mesmo aminoácido. Por exemplo, o aminoácido leucina pode ser codificado pelos códons: CUU, CUC, CUA, CUG, UUA e UUG.
RNA polimerase
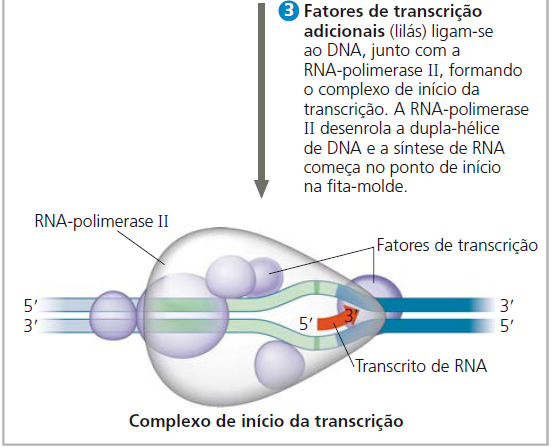
O RNA mensageiro, o carreador da informação do DNA até a maquinaria de síntese de proteínas, é transcrito a partir da fita molde do gene. Uma enzima chamada de RNA polimerase separa as duas fitas de DNA e une os nucleotídeos de RNA complementares à cadeia molde de DNA, aumentando o polinucleotídeo de RNA.
Sequências específicas de nucleotídeos ao longo da cadeia de DNA marcam onde a transcrição de um gene começa e onde termina. A sequência de DNA em que a RNA polimerase se liga e inicia a transcrição é chamada de promotor; em bactérias, a sequência que sinaliza o término da transcrição é chamada de terminador. O mecanismo de término é diferente em eucariotos.
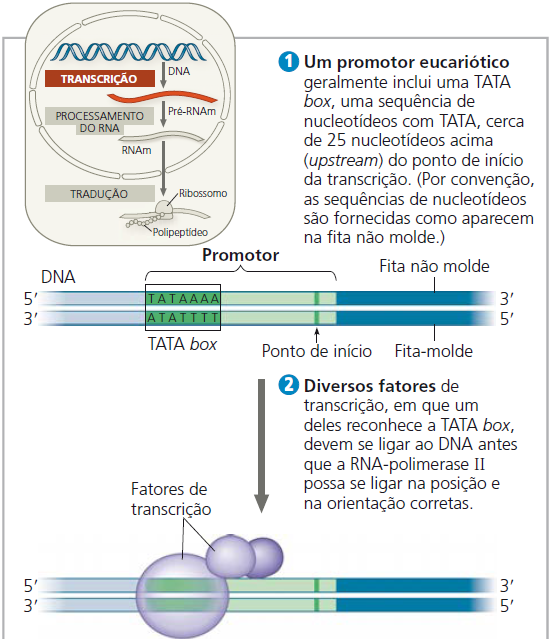
O que é região promotora?
A região promotora é uma sequência específica de nucleotídeos (as “letras” do DNA: A, T, C, G) que fica antes do início de um gene. Essa sequência tem “sinais” que indicam onde a RNA polimerase deve se ligar para iniciar a transcrição do gene.
Um exemplo clássico está no gene da beta globina humana (HBB), que faz parte da hemoglobina. Na região promotora desse gene, encontramos a seguinte sequência:
- 5′ – TATAAA – 3′
Essa é a famosa TATA box, localizada geralmente cerca de 25 a 30 pares de bases antes do início do gene (do códon de início, o ATG). Ela é reconhecida por fatores de transcrição (como o TFIID), que ajudam a posicionar corretamente a RNA polimerase II para começar a transcrição. Vale lembrar que a TATA box não é a única parte da região promotora, existem outras sequências regulatórias.
O que é fita molde e fita codificadora?
O DNA é formado por duas fitas complementares. Durante a transcrição, apenas uma delas é usada pela RNA polimerase como molde para formar o RNA mensageiro (RNAm).
✅ Fita molde (ou fita não codificadora):
- É a fita lida pela RNA polimerase.
- A sequência do RNA é complementar a essa fita.
- Sua direção é 3′ → 5′, pois a RNA polimerase constrói o RNA de 5′ → 3′.
✅ Fita codificadora (ou fita sentido):
- Não é usada como molde, mas tem a mesma sequência do RNAm, exceto que no RNA o T (timina) é substituído por U (uracila).
- Sua direção é 5′ → 3′.
Exemplo:
- Fita codificadora (5′ → 3′): ATG GTC AAC
- Fita molde (3′ → 5′): TAC CAG TTG
- RNA mensageiro (5′ → 3′): AUG GUC AAC
Após a transcrição
No núcleo das células eucarióticas, o RNA recém-formado (chamado de pré-RNAm) passa por modificações antes de sair para o citoplasma. Durante esse processo, conhecido como processamento do RNA, as duas extremidades da molécula são alteradas.
Além disso, em muitos casos, partes internas chamadas íntrons são removidas, e as partes restantes, os éxons, são unidas. Essas mudanças transformam o pré-RNAm em um RNA mensageiro (RNAm) maduro, pronto para ser usado na produção de proteínas.
O que são íntrons e éxons?
O gene não é inteiramente codificável (ou seja, nem todas as suas partes são usadas para formar a proteína). Quando o DNA é transcrito, todo o gene é copiado, incluindo os éxons (partes úteis) e os íntrons (partes não codificantes). Essa transcrição gera o pré-RNAm, que ainda precisa ser processado. Durante o processamento do RNA, os íntrons são removidos e os éxons são emendados para formar o RNAm maduro, que será traduzido em proteína.
- Gene no DNA: [Éxon1] — [Íntron] — [Éxon2] — [Íntron] — [Éxon3]
- Pré-RNAm: Éxon1 + Íntron + Éxon2 + Íntron + Éxon3 ← transcrição completa
- RNAm final: Éxon1 — Éxon2 — Éxon3 ← após remoção dos íntrons
Embora os íntrons não codifiquem proteínas, eles não são inúteis. Eles podem:
- Ajudar na regulação da expressão dos genes;
- Permitir a produção de diferentes proteínas a partir do mesmo gene, por meio do splicing alternativo.
Splicing alternativo
Durante o processamento do pré-RNAm, os íntrons são removidos e os éxons são unidos. No splicing alternativo, nem todos os éxons precisam ser usados sempre na mesma combinação, ou seja, a célula pode escolher quais éxons juntar! Vamos supor que um pré-RNAm tenha 4 éxons:
Éxon 1 — Éxon 2 — Éxon 3 — Éxon 4
A célula pode formar diferentes RNAm maduros assim:
RNAm tipo A:
→ Éxon 1 + Éxon 2 + Éxon 3 + Éxon 4
→ Proteína A
RNAm tipo B:
→ Éxon 1 + Éxon 3 + Éxon 4
→ Proteína B
RNAm tipo C:
→ Éxon 1 + Éxon 2 + Éxon 4
→ Proteína C